Genomweite hochauflösende Chromosomenanalyse mittels Array-CGH
Methode: Agilent 180K Array
Material: EDTA-Blut (3-5 ml) oder > 1µg DNA (Chorionzotten bzw. Fruchtwasserzellen nach Beratung/Absprache)
Dauer: Ca. 4 Wochen
Allgemeine Informationen
- ■ Vor Beginn einer Array-CGH Analyse muss eine Karyotypisierung mittels klassischer Zytogenetik (an Heparinblut) vorausgehen.
- ■ Eine pränatale Array-CGH-Analyse wird nach Rücksprache/Beratung durchgeführt
- ■ Die Kosten einer pränatalen aCGH-Analyse werden in Deutschland zur Zeit noch NICHT von den Krankenkassen übernommen. Wir bieten diese daher als individuelle Gesundheitsleistung (IGeL) an.
- ■ Balancierte strukturelle Aberrationen wie z.B. balancierte Translokationen oder Inversionen, Polyploidien sowie niedriggradige Mosaike werden mit diesem Verfahren in der Regel nicht erfasst.
- ■ Die Analyse erfolgt auf einem 180K Array der Firma Agilent (Auflösung >50 Kilobasen). Die Nomenklatur erfolgt gemäß ISCN2020 (McGowan-Jordan, Hastings, Moore, ISCN2020: An International System for Human Cytogenomic Nomenclature (2020), ISBN: 978-3-318 -06706-4). Die Klassifizierung der Aberrationen erfolgt anhand der ACMG-Kriterien (Riggs et al. 2020; PMID: 31690835).
- ■ Nach Rücksprache besteht die Möglichkeit aCGH+SNP 180K Arrays zu analysieren
Indikationskritierien für eine postnatale Array-CGH-Diagnostik:
- ■ Unklare mentale Retardierung (IQ<70)
- ■ Entwicklungsverzögerung
- ■ Verdacht auf Veränderungen aus dem Bereich der Autismus-Spektrum-Störungen
- ■ Funktionsstörungen bzw. Fehlbildungen des Gehirns
- ■ Multiple dysmorphologischen Auffälligkeiten
- ■ Multiple kongenitale Fehlbildungen
- ■ Wachstumsverzögerungen oder -Auffälligkeiten
Array-CGH als weiterführende Diagnostik:
- ■ Zur genaueren Charakterisierung von Imbalance, die mittels klassischer zytogenetischer Analyse detektiert wurden
- ■ Zum Nachweis fam. Veränderungen (im Zuge einer Segregationsanalyse)
- ■ Als Validierung anderer molekulargenetischen Verfahren (z.B. weiterführende Charakterisierung von MLPA-Befunden)
Indikationskriterien für eine pränatale Array-CGH-Diagnostik:
- ■ Erhöhte Nackentransparenz, auffälliges Ersttrimesterscreening
- ■ Auffälligkeiten in der Nicht-Invasive Pränatal-Testung (NIPT)
- ■ Fetale Auffälligkeiten im Ultraschall
- ■ Zytogenetisch nachgewiesene, nicht näher charakterisierbare Chromosomenveränderung
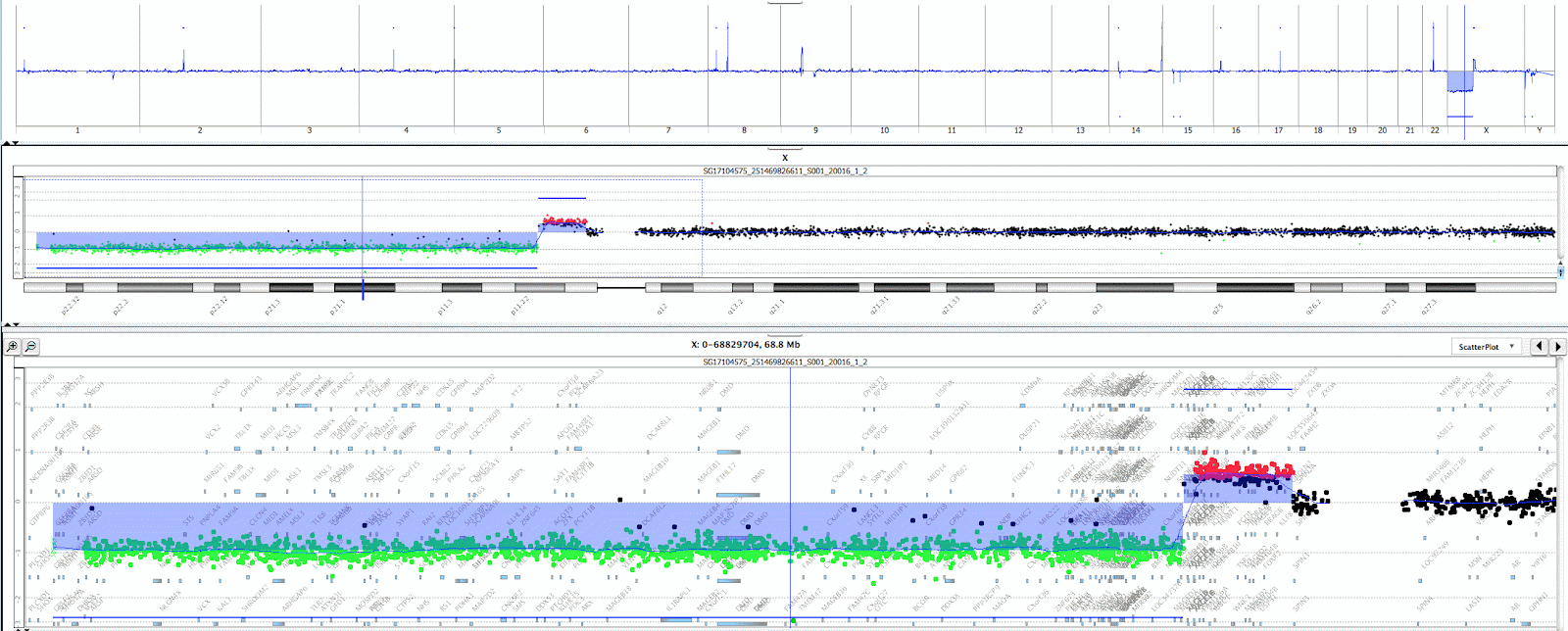
Abb.: Diese Auswertungsansicht in der Array-CGH-Software zeigt eine partielle Monosomie (grün), gefolgt von einer interstitiellen zugewinns (rot) des X-Chromosoms.
